近年来,大语言模型(LLMs)在自然语言处理领域取得了巨大进展,极大地提升了人机交互的效率和精准度。而在计算生物学中,蛋白质序列被类比为自然语言,基于此的蛋白质大语言模型(PLLMs)也应运而生。然而,PLLMs的应用往往需要复杂的预处理和脚本开发,这使得非计算背景的研究人员难以充分利用其潜力。针对这一问题,华中科技大学的研究团队提出了一种名为ProtChat的AI多智能体系统,旨在通过整合GPT-4和多种PLLMs,实现自动化蛋白质分析,降低使用门槛,提升研究效率。
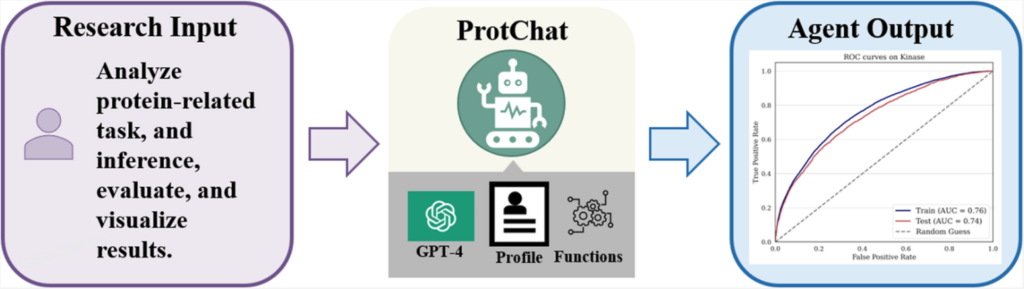
ProtChat是一个基于GPT-4和多种蛋白质大语言模型(如ESM和MASSA)的多智能体系统,旨在简化蛋白质分析流程,降低使用门槛,并为研究人员提供一个高效的自动化工具。
ProtChat的设计与架构
ProtChat 的核心思想是将 GPT-4 的自然语言处理能力与 蛋白质大语言模型(PLLMs) 的蛋白质语义理解能力相结合。通过这种整合,ProtChat 能够自动化执行复杂的蛋白质分析任务,如蛋白质属性预测、蛋白质-药物相互作用预测等。
1. 多智能体架构:分工协作
ProtChat 的系统架构由四个主要智能体组成:
– User Proxy:负责接收用户输入的任务指令,并将其传递给其他智能体。
– Inference:调用PLLMs(如ESM和MASSA)进行蛋白质数据分析,生成推理结果。
– Evaluation:根据推理结果计算任务的相关指标,如准确率、ROC曲线等。
– Visualization:将评估结果可视化,生成图表等输出。
这些智能体通过 Chat Manager 进行协调,动态选择所需的工具并执行任务,整个过程高度自动化。
2. 工作流程:从任务输入到结果可视化
ProtChat 的工作流程通过一个具体的 激酶任务 进行了演示:
1. 用户输入:用户通过 User Proxy 输入任务指令,指定数据文件和任务类型。
2. 推理:Inference 智能体调用PLLM(如MASSA)对蛋白质数据进行分析,生成推理结果。
3. 评估:Evaluation 智能体根据推理结果计算任务的相关指标。
4. 可视化:Visualization 智能体将评估结果可视化,生成图表。
整个过程自动化,用户只需输入指令,系统会自动完成分析、评估和可视化。
ProtChat的应用场景
ProtChat 的应用场景涵盖了蛋白质科学的多个关键领域:
– 蛋白质属性预测:如蛋白质稳定性、荧光强度、远程同源性等。
– 蛋白质-药物相互作用预测:预测蛋白质与药物之间的相互作用,助力药物筛选和设计。
– 蛋白质-蛋白质相互作用预测:预测蛋白质之间的相互作用,揭示复杂的生物网络。
![图片[4]-ProtChat:融合大语言模型与蛋白质语言模型的自动化蛋白质分析工具 --实验盒](../../Attachments/Pasted%20image%2020250120141517.png)
实验结果:自动化与准确性
实验表明,ProtChat 能够自动化处理复杂的蛋白质分析任务,且结果具有较高的准确性。通过与PLLMs(如ESM和MASSA)的集成,ProtChat 在多个下游任务中表现良好,尤其是在蛋白质-药物相互作用和蛋白质-蛋白质相互作用预测方面。
未来研究方向
ProtChat 的成功应用展示了LLMs和PLLMs在蛋白质科学中的潜力,同时也为未来的研究提供了新的思路:
– 指令设计的精确性:为了确保LLMs和PLLMs的有效集成,任务指令的设计需要非常精确,以确保模型能够正确理解和执行任务。
– 基础模型的选择:选择合适的LLM和PLLM基础模型对于任务的成功至关重要。GPT-4 被选为LLM基础模型,而PLLM的选择则取决于任务的具体需求。
– 药物发现中的应用:ProtChat 在药物发现和分子生物学中的应用潜力较大,能够加速新药的开发和分子途径的探索。
代码与数据:开源共享
ProtChat 的代码和数据已公开在 GitHub 上,研究人员可以自由访问和使用:
– GitHub 地址:github.com/SIAT-code/ProtChat
通过开源,ProtChat 为科研社区提供了一个实用的工具,鼓励更多的研究人员参与到这一领域的研究中,推动蛋白质科学的进一步发展。
总结
ProtChat 通过整合GPT-4和蛋白质大语言模型,成功地将自然语言处理能力与蛋白质领域的专业知识相结合,提供了一个高效的自动化蛋白质分析工具。该系统简化了复杂的蛋白质分析流程,并为未来的生物信息学和药物发现研究提供了新的可能性。
随着人工智能技术的不断进步,ProtChat 有望在药物发现、分子生物学等领域发挥更大的作用,推动科学研究的自动化和智能化。
参考文献:
– Huang, H., Shi, X., Lei, H., Hu, F., & Cai, Y. (2025). ProtChat: An AI Multi-Agent for Automated Protein Analysis Leveraging GPT-4 and Protein Language Model. Journal of Chemical Information and Modeling, 65(1), 62-70.
![图片[2]-ProtChat:融合大语言模型与蛋白质语言模型的自动化蛋白质分析工具 --实验盒](https://www.shiyanhe.com/wp-content/uploads/2025/01/Pasted-image-20250120141435-1024x449.png)
![图片[3]-ProtChat:融合大语言模型与蛋白质语言模型的自动化蛋白质分析工具 --实验盒](https://www.shiyanhe.com/wp-content/uploads/2025/01/Pasted-image-20250120141517-1024x397.png)
![图片[5]-ProtChat:融合大语言模型与蛋白质语言模型的自动化蛋白质分析工具 --实验盒](https://www.shiyanhe.com/wp-content/uploads/2021/09/wx-banner-website-2021.png)














暂无评论内容